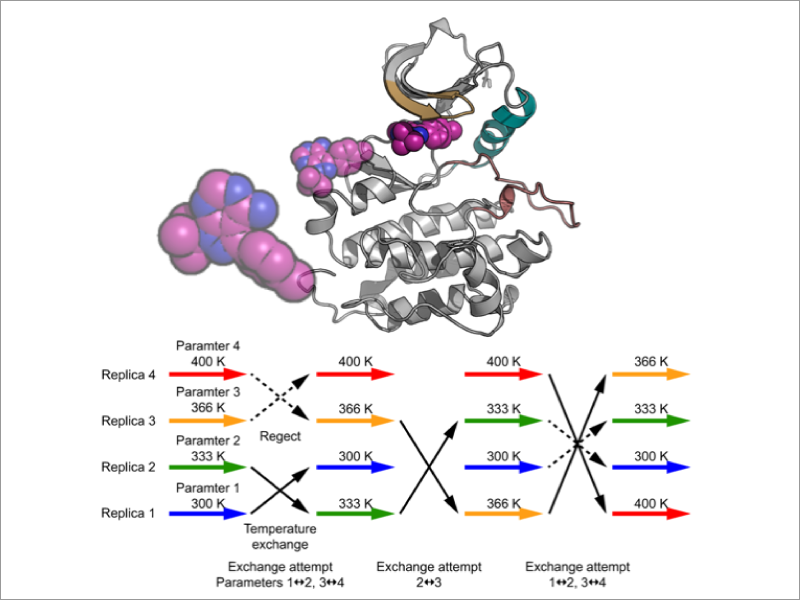
1. 拡張アンサンブル法を用いたタンパク質、リガンドの結合予測
タンパク質による分子認識は、生物科学のみならず創薬分野でも中心的な課題の一つです。古典的には「鍵と鍵穴」モデルで説明され、多くのX線結晶構造に基づいて原子解像度での理解が進みました。一方で、タンパク質やリガンドの構造柔軟性を取り入れた分子認識機構が提案され、理論的にも実験的にも検証が進められています。また、創薬分野では、薬剤化合物の設計の指標として標的タンパク質への結合親和性に加え、滞在時間も考えられる様になっています。分子動力学シミュレーション(MDシミュレーション)は、溶媒効果やタンパク質の構造柔軟性を露わに考慮することで、タンパク質のリガンド結合や会合プロセスを高精度に予測する手段として期待されます。しかし、従来のMDシミュレーションでは、ミリ秒から秒単位を超える時間スケールで起こるリガンド結合、タンパク質会合を直接観測するのは困難です。私たちは、レプリカ交換分子動力学法(REMD法)をはじめとする拡張アンサンブル法を用いた自由エネルギー計算法を開発し、タンパク質―リガンド結合やタンパク質会合プロセスの高精度予測を実現し、タンパク質による分子認識の動的メカニズムの解明を目指します。
参考文献
- Replica-exchange umbrella sampling combined with Gaussian accelerated molecular dynamics for free-energy calculation of biomolecules. Hiraku Oshima, Suyong Re, and Yuji Sugita. J. Chem. Theory Comput., (2019) DOI:https://doi.org/10.1021/acs.jctc.9b00761.
- De Novo Prediction of Binders and Nonbinders for T4 Lysozyme by gREST Simulations. Ai Niitsu, Suyong Re, Hiraku Oshima, Motoshi Kamiya, Yuji Sugita. J. Chem. Inf. Model., 59, 3879-3888 (2019).
- Encounter complexes and hidden poses of kinase-inhibitor binding on the free-energy landscape. Suyong Re, Hiraku Oshima, Kento Kasahara, Motoshi Kamiya, and Yuji Sugita. Proc. Natl. Acad. Sci. USA, 116, 18404-18409 (2019).
- Flexible selection of the solute region in replica exchange with solute tempering: Application to protein-folding simulations. Motoshi Kamiya and Yuji Sugita. J. Chem. Phys., 149, 072304 (2018).
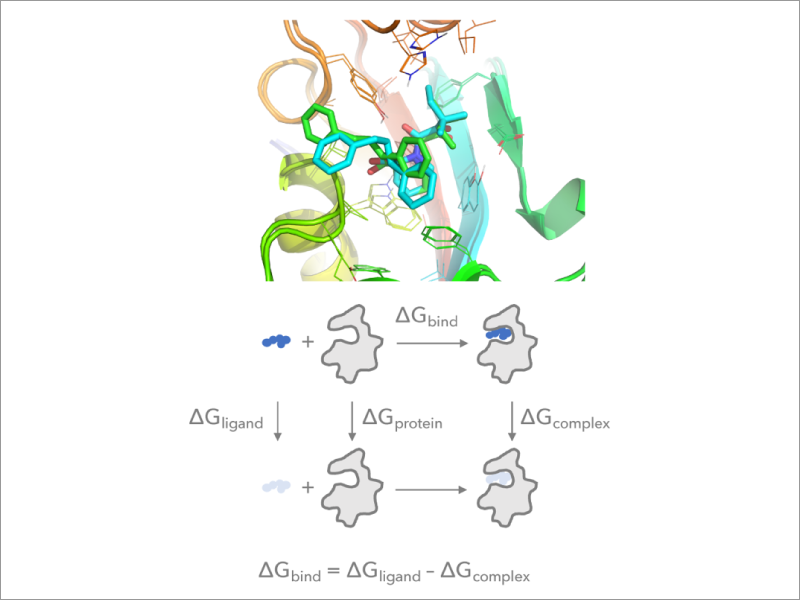
2. 自由エネルギー摂動法のGENESISへの実装
MDシミュレーションを用いた自由エネルギー摂動法(Free Energy Perturbation – FEP法)は、タンパク質―リガンド、タンパク質―タンパク質の会合に伴う自由エネルギー変化(絶対自由エネルギー変化と相対自由エネルギー変化)を予測する最も一般的な手法です。FEP法では、状態間の自由エネルギー変化を多くの中間状態を導入して評価するため計算コストが高くなります。私たちは、スーパーコンピュータの様な超並列計算機を有効に活用した効率的なFEP法を開発しGENESISに実装しました。様々な拡張アンサンブル法と組み合わせ、タンパク質―リガンド結合に限らず、タンパク質の会合やリガンドの膜透過に伴う自由エネルギー変化を高精度・高効率に予測する手法を開発します。